Biotecnologie per l’ambiente
di Flavia Tasso, Chiara Alisi (ENEA - Unità Tecnica Caratterizzazione, Prevenzione e Risanamento Ambientale) Alessia Fiore ENEA (Unità Tecnica Sviluppo Sostenibile ed Innovazione del Sistema Agro-Industriale) Paola Marconi, Salvatore Chiavarini, Carla Ubaldi, Valentina Pinto e Anna Rosa Sprocati (ENEA - Unità Tecnica Caratterizzazione, Prevenzione e Risanamento Ambientale)
Le biotecnologie ambientali hanno un ruolo particolarmente decisivo da svolgere nella realizzazione di uno sviluppo sostenibile duraturo. Alcune esperienze realizzate in ENEA nell’ambito di progetti nazionali ed europei sono rivolte allo sviluppo di approcci innovativi per il monitoraggio a lungo termine dell’impatto ambientale e per il miglioramento delle tecnologie di biorisanamento esistenti. Un’estesa attività di bioprospezione, supportata da tecniche di metagenomica ambientale, è mirata alla cattura e al potenziamento delle capacità di biorisanamento naturalmente presenti nei sistemi ambientali, attraverso la scoperta di nuovi microorganismi, geni funzionali e vie metaboliche, ancora inesplorati
Introduzione
In accordo con le direttive della ricerca Europea in Biotecnologie e in Ambiente, le biotecnologie ambientali includono aspetti di ricerca sia fondamentale, che contribuiscono alla conoscenza dei processi ecologici ed evolutivi, sia orientata verso uno sviluppo tecnologico con esito duplice, economico e ambientale. Di conseguenza, la ricerca biotecnologica ha un ruolo chiave da svolgere nel settore ambientale, ma è importante riconoscere che essa interessa, in misura non trascurabile, anche settori collegati, quali biodiversità, biosicurezza e biotecnologie vegetali. Il dibattito sul rilascio di organismi geneticamente modificati ha catturato per lungo tempo la maggior attenzione, generando una percezione problematica delle biotecnologie, viste come potenziale minaccia per la biodiversità e oscurando gli approcci e le applicazioni biotecnologiche indiscutibilmente compatibili con l’ambiente.
Similmente, anche il mondo microbico - agente chiave delle biotecnologie ambientali- può essere percepito come una potenziale minaccia, data la cattiva fama generale di cui esso gode, conferitagli dall’interesse, per lungo tempo preminente, per i soli microrganismi patogeni di esseri umani, animali e piante. I microrganismi sono, invece, per la maggior parte innocui, anzi utili, anzi indispensabili. In ogni struttura del corpo umano sono presenti batteri indispensabili e in ogni grammo di suolo, ad esempio, troviamo milioni di microrganismi, senza i quali un suolo non potrebbe essere definito tale, per le funzioni che essi svolgono conferendo vitalità: fissazione dell’azoto, mobilizzazione del fosforo, produzione di molecole di utilità per le piante, come fitormoni, siderofori, auxine, vitamine, per citarne alcune. Il ruolo dei microrganismi è principalmente di rendere possibile il riciclo degli elementi in natura, su scala planetaria, restituendoli ai cicli biogeochimici, attraverso i processi di biodegradazione e, in tal senso, possiamo dire che i microrganismi, soprattutto quelli capaci di degradare e detossificare inquinanti tossici, sono certamente cittadini "più verdi" degli esseri umani, che hanno creato situazioni ambientali critiche e caotiche [1].
Tra Europa e Stati Uniti i siti contaminati documentati si aggirano attorno al milione, tra rifiuti pericolosi e contaminanti industriali, con i costi ambientali, sociali ed economici che ne conseguono. Per l’Europa, il problema è oltretutto intensificato per l’elevata densità di popolazione. Tali considerazioni portano a ritenere che le priorità urgenti per garantire uno sviluppo sostenibile durevole debbano includere l’adozione diffusa di tecnologie produttive pulite (processi e prodotti puliti) e di tecnologie per il ripristino degli ecosistemi (biorisanamento ambientale).
In questo quadro di riferimento le biotecnologie hanno un ruolo particolarmente decisivo da svolgere. Raccogliendo alcuni spunti di riflessione [2], grazie alla maturazione delle biotecnologie, è ora possibile prospettare uno scenario più ottimista di quello che Barry Commoner [3] aveva ritratto nel suo libro “The closing circle” sull’incompatibilità della moderna società industriale con la salute ecologica, poiché tra biotecnologia e biodiversità si è stabilito un circuito di flussi sinergici di materiali, servizi e idee. Le biotecnologie ambientali sono un’area della scienza in rapido cambiamento. Al momento, è necessario identificare e verificare le tecnologie-chiave e gli approcci che possano fornire una soluzione alle principali sfide del presente, rappresentate dal cambiamento climatico, dalla diminuzione delle risorse fossili e dalla necessità di migliorare la sostenibilità in agricoltura, nelle attività industriali e domestiche. Per definire lo stato dell’arte e capire come progredire, è utile riferirsi al programma del gruppo di lavoro congiunto US-EU sulle biotecnologie, attivo dal 1990, nato per coordinare gli sforzi transatlantici per promuovere la ricerca sulle biotecnologie e le sue applicazioni orientate al bene della società [4].
Per il periodo 2011-2015 l’obiettivo è arrivare alla comprensione dei processi microbici nei sistemi ambientali d’interesse, promuovendo e sviluppando approcci biotecnologici, che facciano leva sulle ultime tecniche “omics” e le integrino con processi informatici. In questo quadro le principali problematiche di biotecnologie ambientali da affrontare e discutere sono le seguenti: a) sviluppo di approcci che portino un avanzamento nella comprensione della funzione dell’ecosistema microbico in risposta al cambiamento del clima; b) nuovi approcci e nuove idee per processi di biorisanamento e per il monitoraggio a lungo termine dell’impatto ambientale causato dall’inquinamento; c) esplorazione di nuovi approcci informatici per modellare e predire i processi microbici in condizioni ambientali in cambiamento; d) nuovi metodi per valutare la distribuzione spaziale e l’impatto dei processi microbici attraverso scale di osservazione maggiori; e) metagenomica-bioesplorazione ambientale per applicazioni industriali e ambientali (ad esempio ricerca di enzimi estremofili e biocatalizzatori, di nuove specie microbiche e di nuove funzioni metaboliche); f) biotecnologie ambientali applicate al trattamento delle acque reflue, per ridurre la contaminazione delle acque superficiali da parte di inquinanti emergenti e per il recupero di nutrienti e acqua.
Nel presente articolo sono descritte, nelle loro linee generali, alcune attività svolte in ENEA riguardanti le problematiche sopra indicate; per i dettagli scientifici si rimanda alle specifiche pubblicazioni in elenco.
Strategie di risanamento: catturare e mettere a frutto il potenziale di biorisanamento intrinseco dei sistemi ambientali
I processi microbiologici responsabili della trasformazione degli inquinanti (biodegradazione, mobilizzazione, immobilizzazione, detossificazione) rappresentano il motore che guida i fenomeni di attenuazione naturale della contaminazione, cioè la capacità di auto-depurazione degli ecosistemi. Tuttavia l’accumulo di inquinanti altamente tossici in ambiente rende evidente che il flusso dei contaminanti di origine antropica è troppo elevato perché i microrganismi, in condizioni naturali, riescano da soli a smaltirlo. Si rende quindi necessario un aiuto per sfruttare al meglio le loro potenzialità e tradurle in efficaci tecnologie di biorisanamento.
Le vie possibili per attivare e migliorare queste potenzialità naturali comportano un controllo di tipo biogeochimico, che di solito consiste nell’indurre cambiamenti nei parametri fisico-chimici dei sistemi ambientali da trattare (pH, Temperatura, donatori o accettori di elettroni ecc.) o nell’apportare un "aggiustamento di nicchia", mediante adeguati inoculi, che favoriscano la trasformazione degli inquinanti presenti. Un aggiustamento di nicchia può essere realizzato attraverso diversi approcci e le tecniche principali sono la biostimolazione (aggiungere nutrienti per favorire la crescita dei batteri autoctoni) e la bioaugmentation (aggiungere batteri competenti, autoctoni o alloctoni, per aumentare le capacità cataboliche rilevanti per il processo di biorisanamento). Entrambe queste tecniche hanno originato controversie all’interno della comunità scientifica, avendo ognuna sostenitori o detrattori. In realtà un’analisi comparativa delle esperienze condotte in diversi studi indica che non esiste una via univoca, poiché il biorisanamento è una tecnologia sito-specifica e come tale richiede studi dettagliati di caratterizzazione del sito e di fattibilità, prima di decidere sul metodo di biorisanamento più adeguato al caso.
I nostri studi ci hanno portato a sviluppare un approccio al biorisanamento, tramite bioaugmentation, che ci ha permesso di ottenere risultati positivi riguardo alcune problematiche che costituiscono seri ostacoli all’implementazione del biorisanamento, quali la co-contaminazione (concomitanza di metalli pesanti e inquinanti organici) e la bonifica di siti minerari. La logica alla base della bioaugmentation è semplice, tuttavia la tecnica pone diversi problemi di applicazione e ha registrato nel tempo diversi fallimenti [5, 6]. Disparate strategie di bioaugmentation sono state proposte da vari autori, a volte anche concettualmente contrastanti. Lo sfruttamento di “super-specie” [7], l’architettura di biofilm microbici [8], lo sfruttamento della chemiotassi batterica [9], l’inoculo di ceppi contenenti elementi genetici mobili [10], la bioaugmentation della rizosfera [11] sono alcune delle strategie proposte. Un'analisi critica delle sperimentazioni riportate in letteratura evidenzia la generale mancanza di uno scenario ecologico, sia nell'approccio, sia nell'interpretazione dei risultati, che invece é decisivo per un buon esito dell’applicazione. Il nostro lavoro di ricerca è stato quindi orientato verso un’attenta considerazione degli aspetti ecologici sia in fase di scelta dell’inoculo che di interpretazione degli esiti della sperimentazione.
La bioaugmentation è, infatti, influenzata da molteplici fattori, quali le caratteristiche fisico-chimiche dei contaminanti, che possono ridurre l'attività microbica; alcuni elementi che limitano il trasferimento di massa (ad esempio la presenza di argilla o il contenuto di sostanza organica del suolo); fattori di ecologia microbica (flusso energetico, attività microbica spontanea, predatori, competitori, co-substrati, patrimonio genetico dei microrganismi rilevanti, stabilità e attività enzimatica) e metodologia scelta per l’inoculo (selezione dei ceppi, concentrazione e metodi di inoculo, eterogeneità dell'inoculo) [12]. Nel suolo, uno dei principali problemi associati alla bioaugmentation è la sopravvivenza dei microrganismi inoculati o la microbiostasi, cioè l’arresto della crescita e della riproduzione microbica, a causa degli stress abiotici e biotici richiamati in precedenza. La strategia di inoculo è un elemento chiave per poter catturare e mettere a frutto il potenziale microbico spontaneo e diventa quindi una sfida che la ricerca si pone per rendere il biorisanamento una tecnologia matura.
Tra le diverse strategie possibili di bioaugmentation la nostra ricerca è focalizzata sull'uso di consorzi microbici, che mettano in relazione funzionale la struttura della comunità microbica in situ con gli inquinanti presenti. Gli inoculi introdotti, devono essere metabolicamente competenti e in grado di sopravvivere e di catturare il flusso energetico per guidare attivamente l'intera comunità microbica verso i processi biochimici necessari per la bonifica. La procedura da noi individuata per ottenere un inoculo con simili caratteristiche si basa su alcuni criteri razionali: i) le popolazioni microbiche, adattate a situazioni di contaminazione cronica, sono il risultato di una pressione selettiva esercitata dai contaminanti stessi e si possono pertanto considerare popolazioni stabili; ii) la legge di Pareto dice che, all’interno di una comunità, una minima parte degli organismi consuma la gran parte del flusso di energia, per cui serve individuare questa piccola porzione e incrementarla per dirigere il flusso energetico nella direzione desiderata, nello specifico verso una completa rimozione o detossificazione dei contaminanti presenti nel sistema ambientale bersaglio.
Di conseguenza la nostra ricerca è stata indirizzata sulla identificazione delle popolazioni stabili e metabolicamente competenti presenti nelle comunità microbiche autoctone di matrici contaminate, prevalentemente suoli, ma anche sedimenti, acque superficiali e acque reflue. L’approccio seguito comporta che, una volta definito il sistema ambientale da trattare, sia eseguita una caratterizzazione bio-geo-chimica, in particolare vengono definiti: la natura della matrice, la natura dei contaminanti, la loro concentrazione e biodisponibilità (disponibiltà per i sistemi biologici). La comunità microbica nativa viene caratterizzata attraverso un approccio polifasico, sia a livello metabolico che strutturale. L’analisi del profilo metabolico mediante il sistema Biolog-ECOPlates™ fornisce il fingerprint metabolico della comunità (spettro dei substrati che é in grado di utilizzare) e fornisce quindi una misura della diversità funzionale della comunità. L’impiego combinato di tecniche colturali (isolamento della frazione coltivabile, che, come noto, rappresenta una parte molto esigua del totale) e molecolari (cfr. il Focus Metagenomica ambientale) produce informazioni complete sulla comunità microbica, sia a livello strutturale sia ecologico e genetico: composizione in specie, indici di diversità, di ricchezza, di evenness, di organizzazione funzionale, patrimonio metabolico attivo e inducibile, potenziale genetico. L’insieme di queste informazioni permette di valutare il potenziale di biorisanamento intrinseco alle matrici stesse da trattare.
Nel caso in cui il sistema ambientale sia invece privo di microrganismi competenti, quindi si è in assenza di un metabolismo attivo o inducibile verso i contaminanti presenti, è necessario inoculare microrganismi alloctoni, che devono essere scelti sulla base della conoscenza della composizione della comunità nativa, al fine di apportare ceppi compatibili che arricchiscano il patrimonio metabolico della comunità, riducendo al minimo l’impatto sia per gli inoculati che per la comunità nativa [13]. La scelta di procedere con consorzi di batteri, anziché con specie individuali, deriva dall’esigenza di simulare il più possibile i sistemi naturali, dove i microrganismi agiscono in base alle interazioni intra- e inter-specifiche che instaurano, creando complesse reti metaboliche, attraverso le quali arrivano, come intera comunità, alla detossificazione del sistema o alla biodegradazione completa degli inquinanti. Per procedere con la bioaugmentation i ceppi così selezionati vengono cresciuti in laboratorio fino alla concentrazione ritenuta necessaria per effettuare un inoculo di quantità adeguate e successivamente vengono inoculati nella matrice da trattare. Seguono le analisi chimiche ed ecotossicologiche per monitorare il processo e per verificare che non vengano prodotti metaboliti intermedi più tossici dei composti parentali.
Visti da questa prospettiva, i siti contaminati diventano fonte di biodiversità microbica specializzata, che rappresenta una particolare storia evolutiva di adattamento a composti non co-evoluti ma introdotti solo recentemente, in epoca industriale, ex novo (xenobiotici) o in elevate quantità (metalli, petrolio). Partendo da diverse matrici contaminate abbiamo isolato un elevato numero di ceppi batterici di interesse biotecnologico, wild type e non patogeni, che costituiscono la collezione “ENEA-Lilith”. In base alle loro caratteristiche, abbiamo quindi sviluppato alcune formule microbiche adatte a specifici problemi, che verranno brevemente descritti nel seguito: trattamento di acque di conceria, biorisanamento di suoli co-contaminati da metalli e idrocarburi, sviluppo di “tool-boxes“ per il biorisanamento di siti minerari. In accordo con la convenzione sulla biodiversità, tale biodiversità, al pari dei soffioni di acque profonde, dei deserti e di altri ambienti estremi, costituisce un’essenziale risorsa per il nostro futuro e la sua conservazione ex situ è di grande importanza.
Trattamento di reflui conciari.
L’industria conciaria provoca un elevato impatto ambientale a causa dei notevoli quantitativi di effluenti liquidi, solidi e aeriformi prodotti, caratterizzati da elevato contenuto di cromo nei reflui e dei fanghi, elevato livello di COD, presenza di tensioattivi e considerevole salinità negli effluenti. L’ingente consumo di acqua di queste industrie, che utilizzano sia pozzi artesiani sia l’acquedotto pubblico, e la difficoltà di depurare gli effluenti industriali costituiscono i problemi fondamentali dei poli industriali di questo tipo. Le attività svolte (progetto TIDe n.9260, MIUR D.M 593/2000, 2003-2006) riguardano una sperimentazione su impianti pilota, mirata a definire una tecnologia in grado di recuperare la notevole quantità d'acqua utilizzata in questo tipo di lavorazione. Dai fanghi prelevati nelle vasche di decantazione della stessa conceria, è stata isolata e caratterizzata la flora microbica presente, per costituire una formula di ceppi, selezionati per resistenza al Cromo. La formula microbica è stata quindi inoculata a concentrazioni opportune in due impianti pilota che operano con processi diversi: un impianto a fanghi attivi convenzionale (BIO), l’altro con aggiunta di carbone granulare (BAC). Entrambi gli impianti hanno avuto una buona performance, oltre che nella rimozione del Cromo (da 60 a 10-15 mg L-1 in max 70 ore), anche nella completa degradazione degli inquinanti organici presenti (4-cloro-3-metilfenolo, isodecil-polietossilato e paraffine) raggiunta in 40 ore circa e permettendo quindi il riutilizzo dell’acqua effluente. Nell’impianto BAC il carbone attivo ha favorito l’organizzazione della comunità microbica in biofilm, con una più marcata capacità di biodegradazione degli inquinanti e una cinetica di abbattimento del Cromo più rapida (a 15 mg L-1 in 20 ore).
Biorisanamento di suoli co-contaminati da metalli e idrocarburi.
La co-contaminazione rappresenta uno dei colli di bottiglia metabolici del biorisanamento, poiché la tossicità dei metalli inibisce le capacità degradative dei microrganismi, a vari livelli. Partendo da un’esplorazione microbiologica di alcune aree contaminate del sito siderurgico ex-ILVA di Bagnoli, che presentavano differenti tipologie e concentrazioni di inquinanti, sono state isolate tre distinte comunità microbiche native, caratterizzate per la composizione in specie e per il profilo metabolico, per la capacità di resistenza ai metalli pesanti e per la capacità di degradare contaminanti organici, principalmente idrocarburi. È stata inoltre studiata la relazione tra i contaminanti presenti, il livello di ecotossicità rilevato e le caratteristiche metaboliche delle popolazioni microbiche native. Il lavoro, condotto nell’ambito del Progetto TIDe, ha permesso di isolare delle popolazioni stabili, adattate alle condizioni di contaminazione cronica e di selezionare, al loro interno, un centinaio di ceppi batterici con capacità di pluri-resistenza ai metalli pesanti e in grado di mantenere attivo il metabolismo biodegradativo verso idrocarburi e derivati, anche alla presenza di metalli pesanti. Da queste popolazioni, evolutesi per le peculiari condizioni ambientali, rappresentate dalla co-presenza di metalli e inquinanti organici, sono state sviluppate alcune formule microbiche per sperimentazioni di biorisanamento condotte su diverse scale: biometri e microcosmi.
Nella sperimentazione condotta in biometri [14] la formula microbica comprendeva ceppi indigeni di un’area di Bagnoli, appartenenti ai generi Arthrobacter, Bacillus, Delftia, Exiguobacterium, Pseudomonas e Rhodococcus ed è stata impiegata come agente di bioaugmentation per studiare la fattibilità di biorisanamento di un suolo co-contaminato da gasolio (1% w/v) e metalli pesanti. In 42 giorni di incubazione a 28 °C gli idrocarburi lineari n-C12-20 sono stati completamente degradati, mentre gli isoprenoidi sono stati ridotti del 60%. I test ecotossicologici seguiti hanno evidenziato una netta riduzione della tossicità, legata al tempo di trattamento. Mediante la tecnica molecolare t-RFLP (terminal-Restriction Fragment Lenght Polymorphism) è stata studiata la sopravvivenza della formula microbica impiegata, rilevando sia frammenti corrispondenti a ceppi inoculati di Pseudomonas, Arthrobacter, Exiguobacterium e Delftia che frammenti non ascrivibili alle specie inoculate, suggerendo che si sia instaurato un equilibrio funzionale tra i microrganismi introdotti e quelli nativi.
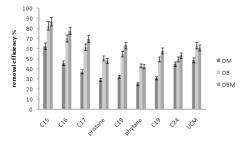
La ricerca è proseguita con uno scale up del sistema sperimentale, realizzato mediante l’impiego di microcosmi terrestri (ASTM 2004 Guidelines E-1197), sistemi indisturbati adatti a riprodurre condizioni sperimentali il più possibile vicini a quelli in campo, in quanto preservano intatta la complessità ecologica del suolo, su scala microbiologica e biochimica. Le informazioni ottenute nei microcosmi sono ritenute estrapolabili direttamente al campo, a differenza di quelle ottenute in sistemi sperimentali troppo semplificati perché possano essere ritenuti significativi dell’ecosistema target. La formula microbica usata in precedenza è stata potenziata, con l’aggiunta di ceppi produttori di biosurfattanti, sempre isolati dal sito di Bagnoli, allo scopo di aumentare la biodisponibilità degli idrocarburi. Lo studio ha dimostrato che la formula microbica introdotta è in grado di promuovere una degradazione degli idrocarburi prossima al 75% nei primi 15 cm di suolo argilloso, in presenza di metalli pesanti (Zinco 500 mg kg-1 di suolo e Piombo 1000 mg kg-1 di suolo), come mostrato in figura 1. Lo studio della mobilità dei metalli pesanti ha dimostrato che, al termine dell’esperimento, essi sono presenti in forma quasi completamente biodisponibile. L’analisi del profilo molecolare dell’rDNA 16S delle comunità microbiche, eseguita attraverso la tecnica della DGGE e la successiva applicazione degli indici ecologici [15], ha mostrato che in tutte le condizioni sperimentali testate viene preservata un’alta ricchezza in specie (Indici Rr e 1-D) e non si osservano significativi cambiamenti all’interno della struttura delle comunità, mentre si osserva una specializzazione dove avviene la biodegradazione. Il confronto dei dati ottenuti dalle analisi metaboliche (sistema Biolog) e molecolari (DGGE) mette in luce il ruolo positivo svolto dalla formula sia nell’attivare il processo di biodegradazione che nel guidare l’intera comunità microbica, indigena e inoculata, verso la biodegradazione dei contaminanti.
Biorisanamento di siti minerari.
Nell’Unione Europea, la superficie di suoli influenzati da attività minerarie è stata stimata pari allo 0,6%, rispetto a una media mondiale dello 0,2%. La bonifica dei suoli contaminati da metalli pesanti che derivano da attività minerarie rappresenta, quindi, un obiettivo strategico per le politiche europee. Il progetto UMBRELLA (FP7, Grant No. 226870, 2009-2012), cui partecipa la nostra Unità, mira a sviluppare un approccio al biorisanamento, che permetta di stabilire misure migliorative, economicamente efficienti e sostenibili, per la bonifica di siti contaminati da metalli pesanti, in diverse regioni geografiche e climatiche d’Europa, su larga scala. L'obiettivo generale del progetto è di utilizzare il potenziale offerto dai microrganismi per accelerare le attuali tecniche di fitorisanamento. L’obiettivo viene perseguito con un approccio di ricerca interdisciplinare nei settori della microbiologia, dell’assorbimento di metalli da parte di piante e della (idro)-geochimica, mirato allo studio dell'influenza microbica sui cicli biogeochimici dei metalli e dell’impatto del loro impiego nella protezione del suolo e delle acque. Il progetto si propone di fornire agli utilizzatori finali della tecnologia un insieme di strumenti (microrganismi e piante in associazione, approccio metodologico e modelli previsionali) per azioni di bonifica in situ, a basso costo e a basso impatto ambientale. Al progetto partecipano Università, Istituzioni scientifiche, SMEs e Agenzie governative per fornire ai governi linee guida uniformi per la protezione del suolo e dell'acqua, in modo da superare le pratiche separate delle diverse Agenzie, come avviene ora. Il progetto ha preso in esame complessivamente sei siti minerari in diverse zone europee, secondo un unico approccio modellistico, per garantire la futura applicabilità in tutta Europa.
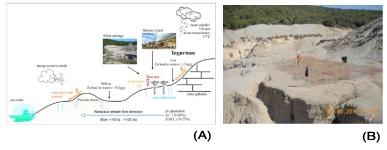
Il contributo delle biotecnologie microbiche al progetto consiste nell’individuazione dei microrganismi autoctoni dei suoli delle sei miniere e la successiva costituzione di un consorzio microbico, per ogni sito, con i 10 ceppi batterici selezionati per le migliori capacità di resistenza ai metalli pesanti e di promozione di crescita delle piante (PGP). I consorzi microbici così definiti entrano a far parte di un “tool-box” specifico per ogni sito, insieme alle specie botaniche selezionate, preferibilmente endemiche, per realizzare interventi di fitorisanamento assistito di metalli pesanti (fitoestrazione o fitoimmobilizzazione). Il sito italiano prescelto è la miniera abbandonata di blenda e galena (Zn e Pb) di Ingurtosu, Sardegna. Nel sito è stato allestito un campo sperimentale (figura 2), dove sono tuttora in corso dei test che impiegano come tool-box un’associazione tra una pianta pioniera endemica, Euphorbia pythiusa, un consorzio di batteri PGP (UI) e, in alcune condizioni, anche micorrize autoctone. È stato inoltre testato l’effetto di un prodotto commerciale (ViroMine™) sul dilavamento dei metalli pesanti dal suolo. L’attività microbica del suolo, i parametri fisiologici delle piante, il contenuto di metalli pesanti nel suolo e nelle piante sono stati monitorati durante la prova, mostrando una certa capacità spontanea di assorbimento di zinco da parte della pianta, un effetto positivo dei batteri sullo stato di salute della pianta e una riduzione della concentrazione dei metalli nel suolo, particolarmente significativa nelle condizioni in cui sono presenti sia batteri che ViroMine. L’elaborazione dei dati sperimentali è in corso e sarà oggetto di pubblicazioni scientifiche coordinate tra i vari partner. Per ogni sito è stato definito un modello di percorsi di scarico e un modello previsionale di dispersione dei metalli. In figura 2 è riportato il modello del sito italiano di Ingurtosu. Informazioni disponibili sul sito web http://www.umbrella.uni-jena.de/cms/index.php
Nota conclusiva
Le attività descritte sono orientate a individuare strategie e approcci di biorisanamento che contribuiscano a rendere la tecnologia maggiormente fattibile, stabile e matura. Allo stesso tempo contribuiscono ad allargare le conoscenze fondamentali sulla biodiversità microbica, sui processi metabolici e sulle interazioni tra microrganismi e tra microrganismi e piante, in ecosistemi sottoposti a forte stress ambientale. L’approccio alla bioaugmentation proposto, che si avvale di consorzi microbici selezionati secondo criteri di ecologia microbica, è stato applicato con successo in diverse situazioni sperimentali di contaminazione e in diverse matrici, confermando che una selezione razionale dell’inoculo microbico, operata tenendo conto del contesto ecologico in cui si deve agire, può effettivamente contribuire a catturare e mettere a frutto il potenziale di biorisanamento intrinseco dei sistemi ambientali contaminati. Lo sviluppo della metagenomica ambientale ha posto le basi disciplinari e tecniche per amplificare enormemente la capacità di bioprospezione e nei prossimi anni sarà possibile rilevare in modo più realistico questo potenziale metabolico, con la scoperta di nuovi organismi, geni funzionali e vie metaboliche ancora sconosciute. Da tutto questo trarranno vantaggio sia le tecnologie di biorisanamento, sia le tecniche di monitoraggio ambientale. Soluzioni efficaci per le problematiche di risanamento trattate, quali la co-contaminazione e la bonifica di siti minerari, o per problematiche nuove, come gli inquinanti emergenti, rappresentano obiettivi strategici per l’Europa.
Ringraziamenti
Le attività descritte sono state condotte con il contributo di numerosi ricercatori, citati nelle pubblicazioni. Si ringraziano i partner del consorzio UMBRELLA e in particolare il dottor Giovanni De Giudici, dell’Università di Cagliari, per averci introdotto nel mondo minerario della Sardegna, svelandocene con passione e competenza gli aspetti geologici e storici.
Bibliografia
- J. Ramos (2000), EC-US taskforce on biotechnology research, European Communities, ISBN 92-894-0110-9.
- A.T. Bull (1996), “Biotechnology for environmental quality: closing the circles”, Biodiversity and Conservation, volume 5, pages 1-25.
- B. Commoner (1971), The closing circle, Bantam Edition, New York, ISBN 0-553-12921-X
- http://ec.europa.eu/research/biotechnology/eu-us-task-force/index_en.cfm.
- M. Bouchez, D. Blanchet, V. Bardin, F. Haeseler, J.P. Vandecasteele (1999), “Efficiency of defined strains and of soil consortia in the biodegradation of polycyclic aromatic hydrocarbon (PAH) mixtures”, Biodegradation, volume 10(6), pages 429-35.
- S.Z. Fu, H.X. Fan, S.J. Liu, Y. Liu, Z.P. Liu (2009), “A bioaugmentation failure caused by phage infection and weak biofilm formation ability”, Journal of Environmental Science, volume 21, pages 1151-1161.
- A.C. Singer, C.J. van der Gast, I.P. Thompson (2005), “Perspectives and vision for strain selection in bioaugmentation”, Trends in Biotechnology, volume 23(2), pages 74-77.
- S. Wuertz, S. Okabe, M. Hausner (2004), “Microbial communities and their interactions in biofilm systems: an overview”, Water Science and Technology, volume 49 (11-12), pages 327-336.
- S. Venkata Mohan, T. Kisa, T. Ohkuma, R.A. Kanaly, Y. Shimizu (2006), “Bioremediation technology for treatment of PAH-contaminated soil and strategies to enhance process efficiency”, Reviews in Environmental Science and Biotechnology, volume5, pages 347-374.
- I.P. Thompson, C.J. van der Gast, Singer A.C. (2005), “Bioaugmentation for bioremediation: the challenge of strain selection, Environmental Microbiology, volume 7 (7), pages 909-915.
- T.J. Gentry, C. Rensing, I.L. Pepper (2004), “New approaches for bioaugmentation as a remediation technology”, Critical Reviews in Environmental Science and Technology, volume 34, pages 447-494.
- T.M. Vogel (1996), “Bioaugmentation as a soil bioremediation approach”, Current Opinion in Biotechnology, volume 7(3), pages 311-316.
- A.R. Sprocati, C. Alisi, F. Tasso, P.Marconi, A. Sciullo, V. Pinto, S. Chiavarini, C. Ubaldi and C. Cremisini ((2012), “Feasibility study for bioremediation of a soil co-contaminated by diesel oil and heavy metals using a tailor-made microbial formula as bioaugmentation agent”, Process Biochemistry, 47:1649–1655.
- C. Alisi, R. Musella, F. Tasso, C. Ubaldi, S. Manzo, C. Cremisini and A.R. Sprocati (2009), “Bioremediation of diesel oil in a co-contaminated soil by bioaugmentation with a microbial formula tailored with native strains selected for heavy metals resistance”, Science of the Total Environment, volume 407 (8), pages 3024-3032.
- M. Marzorati, L. Wittebolle, N. Boon, D. Daffonchio, W. Verstraete (2008), “How to get more out of molecular fingerprints: practical tools for microbial ecology”, Environmental Microbiology, volume 10(6), pages 1571–1581.
- P.J. Turnbaugh and J.I. Gordon (2008), “An invitation to the marriage of metagenomics and metabolomics”, Cell, volume 134, pages 708–713.
- G.W. Tyson, J. Chapman, P. Hugenholtz, E.E. Allen, R.J. Ram, P.M. Richardson, V.V. Solovyev, E.M. Rubin, D.S. Rokhsar, J.F. Banfieldm (2004) “Community structure and metabolism through reconstruction of microbial genomes from the environment”, Nature, volume 428(6978), pages 37-43.
- J.C. Venter et al. (2004), “Environmental genome shotgun sequencing of the Sargasso Sea”, Science, volume 304(5667), pages 66-74.
- S. Mirete, C.G. de Figueras, J.E. González-Pastor (2007), “Novel nickel resistance genes from the rhizosphere metagenome of plants adapted to acid mine drainage”, Applied and Environmental Microbiology, volume 73 (19), pages 60001-60011.
- J.R. Marchesi, A.J. Weightman (2003), “Comparing the dehalogenase gene pool in cultivated alpha-halocarboxylic acid-degrading bacteria with the environmental metagene pool”, Applied Environmental Microbiology, volume 69(8), pages 4375-4382.
- C. Simon and R. Daniel (2011), “Metagenomic Analyses: Past and Future”, Trends in Applied and Environmental Microbiology, volume 77, pages 1153–1161.
- E. Yergeau, S. Sanschagrin, D. Beaumier, C. Greer (2012), “Metagenomic analysis of the bioremediation of diesel-contaminated Canadian high artic soils”, PLoS One, volume 7(1), e30058, doi 10.1371/journal.pone.0030058.
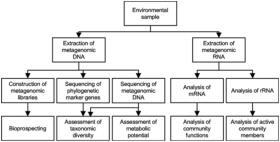
Focus: Metagenomica ambientale
Si stima che le cellule microbiche presenti sul pianeta siano nell’ordine di 1030 [16] e il numero di specie nell’ordine di 106-108. È noto che oltre il 99,8% dei microrganismi presenti nei diversi ambienti naturali è incoltivabile con le metodiche in uso e quindi inaccessibile per eventuali approcci biotecnologici. L’avvento delle tecniche “metagenomiche” -indipendenti dalla coltivazione- ha permesso di rilevare l’esistenza di specie batteriche finora sconosciute, i cui genomi codificano per un serbatoio di prodotti e funzioni, in gran parte inutilizzato. La metagenomica diventa quindi un potente strumento d’indagine per l’esplorazione dell’ecologia di complesse comunità microbiche e del loro potenziale metabolico, in una grande varietà di ambienti, con una considerevole ricaduta sullo sviluppo delle biotecnologie ambientali e sui settori a esse collegati, in particolare la biodiversità. Gli approcci alle analisi metagenomiche sono essenzialmente due: l’analisi strutturale e l’analisi funzionale che, combinate, producono un enorme impatto sulla conoscenza dell’intera comunità microbica e sulle dinamiche di popolazione [17, 18], sulla scoperta di nuovi geni che esprimono tratti caratteristici, come ad esempio geni di resistenza ad antibiotici, a metalli pesanti [19] o attività enzimatiche specifiche del metabolismo primario o secondario [20] [cfr.diagramma di flusso, [21]]
In tal modo si può valutare il potenziale metabolico dell’intera comunità, anche miratamente ad applicazioni specifiche.
Uno dei lavori pionieri di metagenomica ambientale è stato eseguito analizzando il genoma di un drenaggio acido di miniera in California, uno degli ambienti più estremi al mondo [17]. In questo ambiente la comunità microbica forma un biofilm rosa che galleggia sulla superficie dell’acqua.
L’acqua sottostante ha un valore di pH intorno a 1, un’elevata concentrazione di metalli pesanti (Fe, Zn) e assenza di forme di carbonio o azoto assimilabili, tranne la forma gassosa. Attraverso la ricostruzione di interi genomi microbici è stata determinata la struttura della comunità microbica ed è stato ricostruito un modello biogeochimico, individuando le vie metaboliche per il carbonio, per la fissazione dell'azoto e per la produzione di energia e ampliando le conoscenze sulle strategie di sopravvivenza in ambienti estremi. Si può quindi comprendere come la metagenomica, applicata ad ambienti estremi o suoli contaminati, trovi larga applicazione nell’individuazione di quei microrganismi o geni attivi nella degradazione di contaminanti ambientali [22].
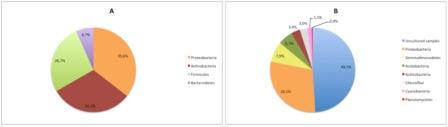
Il nostro laboratorio, nell’ambito del progetto “Umbrella”, ha applicato tecniche di metagenomica, accanto alle tecniche colturali, allo scopo di esplorare la biodiversità batterica in campioni di suolo della ex miniera di Ingurtosu (Sardegna), contaminati da metalli pesanti (Zn e Pb). È stata eseguita un’analisi filogenetica sul rDNA 16S, costruendo due librerie clonali dal DNA totale presente. L’analisi delle sequenze derivate dalle due librerie (174 cloni per gli Archaea e 258 cloni per gli Eubatteri) ha evidenziando una ricchezza di biodiversità inaspettata in una matrice tanto ostile (scarti di miniera), priva di nutrienti, con scarsissima materia organica e con elevatissime concentrazioni di Pb e Zn in forma biodisponibile. In particolare la maggior parte dei cloni individuati per gli Eubatteri, non era stata rilevata neppure a livello di phylum mediante le tecniche colturali (Cianobatteri, Cloroflexi, Gemmatimonadates) (figura 3). Il lavoro procede per attingere in questo serbatoio metabolico inesplorato a funzioni target di interesse per le applicazioni di biorisanamento in corso sul sito (fitorisanamento assistito con batteri), in particolare funzioni di promozione di crescita delle piante e regolazione della mobilità dei metalli. Un obiettivo a più lungo temine è l’individuazione di microorganismi e funzioni metaboliche di interesse in tematiche bio-geo-chimiche aperte, come la completa comprensione del ruolo dei microorganismi nella formazione dei suoli, che rimane un problema ancora irrisolto in biologia.